× schließen
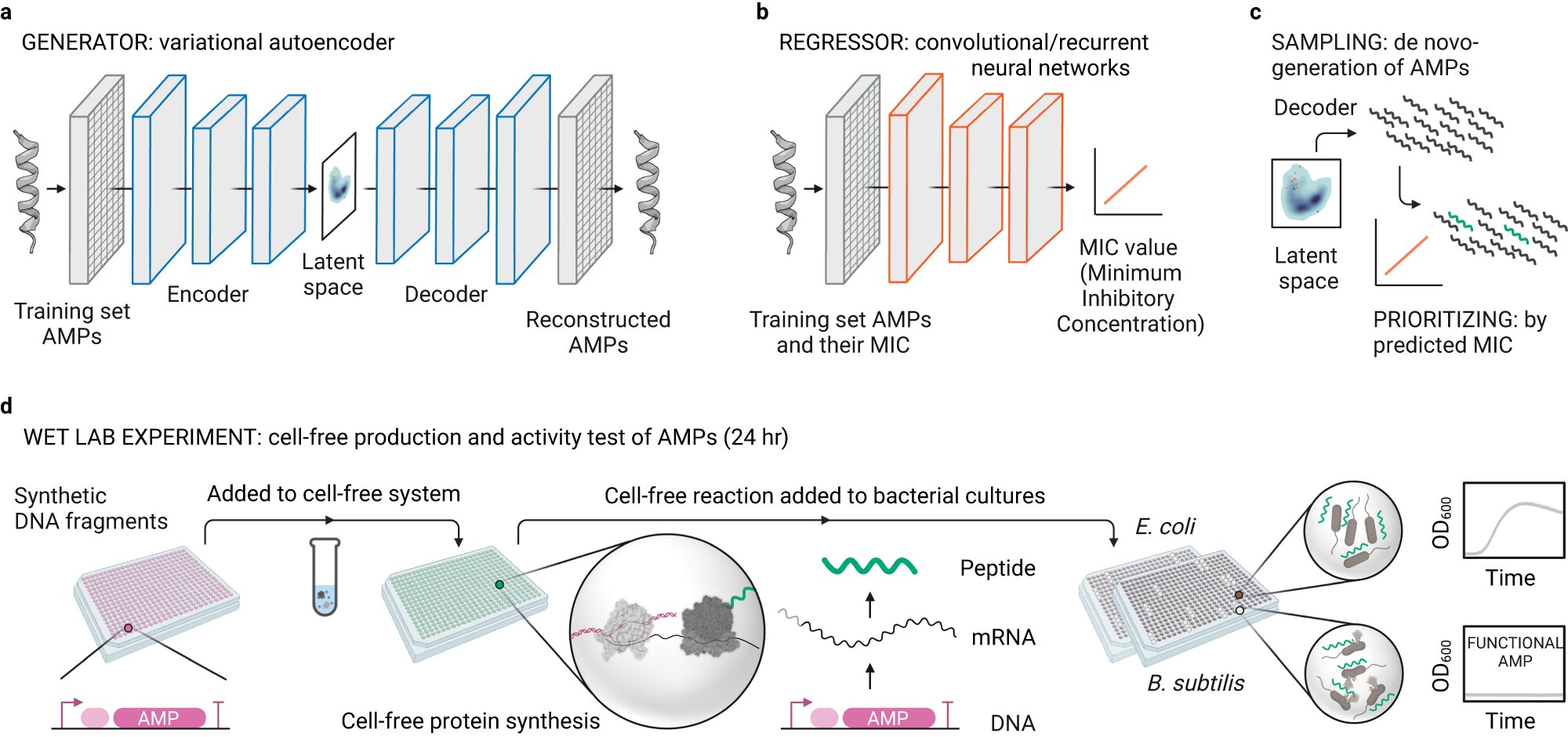
Der Arbeitsablauf für die De-novo-Entwicklung von AMPs mittels Deep Learning und zellfreier Biosynthese. a Generative Variations-Autoencoder (VAEs) für das De-novo-AMP-Design nach dem Training mit bekannten AMP-Sequenzen. b Faltungs- oder rekurrente Feedforward-Neuronale Netze als Regressoren für die MIC-Vorhersage, nachdem sie auf bekannten AMPs und ihren MICs trainiert wurden. c Trainierte generative und prädiktive Modelle werden für die Stichprobenentnahme aus dem latenten Raum (De-novo-Design von AMPs) bzw. für die Priorisierung von AMPs (Vorhersage ihres MIC) verwendet. d Experimentelle Pipeline für die schnelle zellfreie Biosynthese von AMPs, die aus synthetischen DNA-Fragmenten entwickelt wurde, und direktes Testen der in der zellfreien Mischung produzierten AMPs an Bakterienkulturen, gefolgt von einem kontinuierlichen Wachstumstest über Nacht. Erstellt mit BioRender.com. Kredit: Natürliche Kommunikation (2023). DOI: 10.1038/s41467-023-42434-9
Die zunehmende Resistenz von Bakterien gegen Antibiotika stellt ein wachsendes globales Gesundheitsrisiko dar. Nun haben Forscher am Max-Planck-Institut für terrestrische Mikrobiologie in Marburg, Deutschland, synthetische Biologie und künstliche Intelligenz (KI) kombiniert, um einen effizienteren Ansatz zur Suche und Herstellung neuer antimikrobieller Peptide zu entwickeln, die gegen eine Vielzahl von Bakterien wirksam sind. Ihr Papier ist veröffentlicht im Magazin Natürliche Kommunikation.
Bioaktive Peptide spielen eine Schlüsselrolle in Gesundheit und Medizin. Derzeit werden mehr als 80 Peptidmedikamente verwendet, die alle aus natürlichen Quellen isoliert werden. Es wird jedoch geschätzt, dass Antibiotikaresistenzen jedes Jahr weltweit zu mehr als einer Million Todesfällen führen. Es wird erwartet, dass diese Zahl bis 2050 10 Millionen erreichen wird, wodurch ein dringender Bedarf an neuen Methoden entsteht, um die Entwicklung neuer antimikrobieller Mittel zu beschleunigen. Ungenutztes Potenzial liegt im unnatürlichen Raum, wo schätzungsweise 2.010 bis 2.030 verschiedene Peptide noch erforscht werden müssen.
Pipeline für neuartige bioaktive Peptide
In Zusammenarbeit mit mehreren Laboren des Max-Planck-Instituts für terrestrische Mikrobiologie, der Universität Marburg, dem Max-Planck-Institut für Biophysik, dem Institut für Mikrobiologie der Bundeswehr, dem iLung-Institut und dem INRAe Frankreich leitete ein Team von Wissenschaftlern des Max-Planck-Instituts von Tobias Erb hat eine neue Pipeline für die Entwicklung bioaktiver Peptide aufgebaut.
„Beim Deep Learning lernt ein neuronales Netzwerk (vom menschlichen Gehirn inspirierte Algorithmen) aus großen Datenmengen. Diese Art des maschinellen Lernens ist vielversprechend für die Peptidentdeckung und das De-novo-Design. Normalerweise folgt jedoch die chemische Synthese von Peptiden.“ für die experimentelle Validierung, was ziemlich schwierig und zeitaufwändig ist und die Anzahl der Peptide, die chemisch synthetisiert werden können, erheblich einschränkt“, erklärt Amir Pandi, Hauptautor der Studie.
Um diese Einschränkungen zu überwinden, richtete das Forschungsteam eine zellfreie Proteinsynthese-Pipeline für die schnelle und kostengünstige Produktion antimikrobieller Peptide direkt aus DNA-Vorlagen ein. Das neue Protokoll bietet eine schnelle, kostengünstige Hochdurchsatzmethode zum Screening antimikrobieller Peptide.
Das Team nutzte zunächst generatives Deep Learning, um Tausende von antimikrobiellen Peptiden de novo zu entwickeln, und grenzte sie dann durch prädiktives Deep Learning auf 500 Kandidaten ein. Davon identifizierte das Screening mit der zellfreien Pipeline 30 funktionelle Peptide, die die Forscher dann durch Molekulardynamiksimulationen, antimikrobielle Aktivität und Toxizität charakterisierten.
Breitbandwirkung gegen Krankheitserreger
Bemerkenswerterweise zeigten sechs der De-novo-Peptide eine Breitbandaktivität gegen multiresistente Krankheitserreger und entwickelten keine bakterielle Resistenz. „Wir haben von dieser Kombination aus zellfreier synthetischer Biologie, künstlicher Intelligenz und einem Hochdurchsatzansatz stark profitiert. Durch die Erhöhung der Zahl der Kandidaten, die in weniger als 24 Stunden experimentell getestet werden können, sind die Chancen, aktive antimikrobielle Peptide zu finden, gestiegen. „, sagt Pandi.
„Unsere zellfreie Proteinsynthese-Pipeline ergänzt somit nicht nur die jüngsten Fortschritte im computergestützten Design. Sie hat auch das Potenzial, die Beziehung zwischen Design und Funktion bioaktiver Peptide schneller und kostengünstiger zu untersuchen.“
Tobias Erb fügt hinzu: „Diese neue Methode an der Schnittstelle von synthetischer Biologie und maschinellem Lernen wird für Wissenschaftler interessant sein, die in den Bereichen Biomedizin und bioaktive Peptide tätig sind.“ »
Zu den nächsten Schritten gehören die weitere Verbesserung der Peptidproduktionsausbeute sowie die Verwendung von KI- und synthetischen Biologie-Ansätzen zur Entwicklung neuer antimikrobieller Peptide, die stabiler und weniger toxisch sind, oder die Hinzufügung einer spezifischen Wirkungsweise. Die Forscher planen außerdem die Anwendung erweiterter tiefer generativer Modelle, bei denen die Maschine molekulare Darstellungen gewünschter Eigenschaften lernt, was die Erfolgsquote bei der Identifizierung aktiver Kandidaten verbessern würde.
Mehr Informationen:
Amir Pandi et al.: Zellfreie Biosynthese in Kombination mit Deep Learning beschleunigt die De-novo-Entwicklung antimikrobieller Peptide. Natürliche Kommunikation (2023). DOI: 10.1038/s41467-023-42434-9

„Freiberuflicher Kommunikator. Hardcore-Web-Praktiker. Unternehmer. Totaler Student. Bier-Ninja.“